2 回答
TA贡献1848条经验 获得超2个赞
首先,你提到在回归分类中预测新基因的疾病评分,你是什么意思?输出似乎是二进制的,0或1,因此这是一个二进制分类问题。您应该改用xgboost's 分类器。更新:让我们根据评论假设一个回归问题来模拟您的情况。尽管对于下面的示例,我们应该设置'objective':'multi:softmax'为输出实际标签。
根据您的问题,您似乎要做的就是在那些未正确预测的样本上索引测试集,并分析误导性的特征,这具有一定的意义。
让我们用一些示例数据集重现您的问题:
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
import shap
import xgboost
X,y = shap.datasets.iris()
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3)
model = xgboost.train(params={"learning_rate": 0.01},
dtrain=xgboost.DMatrix(X_train, label=y_train),
num_boost_round =100)
使用整个测试集的 SHAP 图非常简单。举个force_plot例子:
explainer = shap.TreeExplainer(model)
shap_values = explainer.shap_values(X_test)
shap.initjs()
shap.force_plot(explainer.expected_value, shap_values, X_test)
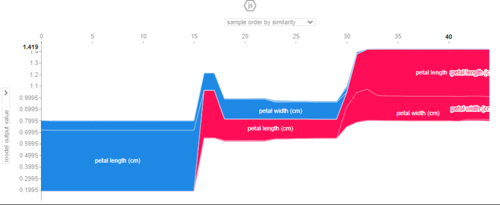
现在,如果我们想对错误分类的样本执行相同的操作,我们需要查看输出概率。由于 iris 数据集有多个类,假设我们想要可视化force_plot那些应该分类为 的样本2,但我们有一个输出值如下1.7:
y_pred = model.predict(xgboost.DMatrix(X_test))
m = (y_pred <= 1.7) & (y_test == 2)
现在我们使用掩码对集合执行布尔索引X_test,并更新shap_values:
shap.initjs()
c= explainer.shap_values(X_test[m])
shap.force_plot(explainer.expected_value, shap_values, X_test[m])
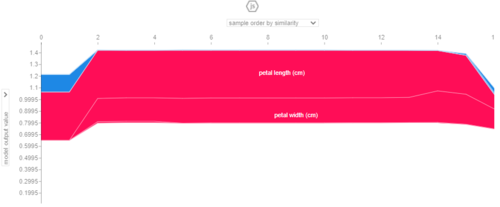
这告诉我们,花瓣的长度和宽度主要将回归推向更高的值。因此,它们可能是在错误分类中发挥主要作用的变量。
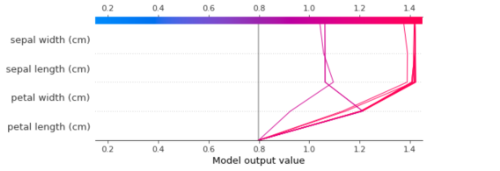
同样,对于一个decision_plot:
shap.decision_plot(explainer.expected_value, shap_values,
X_test[m], feature_order='hclust',
return_objects=True)
TA贡献1820条经验 获得超10个赞
由于我没有您的数据集,因此无法检查代码,但这里的一些想法可能会为您指明方向。
看来你没有训练你的回归者。应该是像线一样
xgbr = xgboost.XGBRegressor()
xgbr.train(X, Y)
现在你可以使用了xgbr.predict(X);)
您还需要培训解释员:
explainer = shap.TreeExplainer(xgbr)
with warnings.catch_warnings():
warnings.simplefilter("ignore")
sh = explainer.shap_values(X)
现在您可以选择值:
misclassified = (y_pred <= 0.7) & (Y == 1)
shap.decision_plot(expected_value, sh, features_display, link='logit', highlight=misclassified)
在使用之前,shap我建议您检查回归器对数据的拟合程度。因此,为此我建议您将部分数据用于测试,而不是在训练中使用它。然后,您可以通过计算和比较测试集和训练集的 MSE 来评估拟合优度。差异越大,预测器的表现就越差。
添加回答
举报
