2 回答
TA贡献1848条经验 获得超6个赞
除了您对情节的积极评论之外,这里是一个如何为数据集实现多轴的示例。
该代码比看起来简单得多。由于我格式化dicts 以便于阅读的方式,代码显得“冗长”。
关键要素是:
time添加列 ( )的累积和time_c以供使用xaxis2。添加与 对齐的隐藏跟踪
xaxis,以及与 对齐的时间数据xaxis2。如果没有隐藏迹线,则两个轴要么不出现,要么出现但未对齐,因为只绘制了一条迹线。
(更新)示例代码:
以下代码已更新,以解决 OP 在使用更大(70k 行)数据集时遇到的问题。
layout['xaxis']键的更改是对和字典的更新,layout['xaxis2']以包含'type': 'category','nticks'和 定义的'range'键。
import pandas as pd
from plotly.offline import plot
# Create the dataset.
raw_data = {'time': [21.9235, 4.17876, 4.02168, 3.81504, 4.2972],
'tpu': [33.3, 33.3, 33.3, 33.3, 33.3],
'cpu': [32, 32, 32, 32, 32],
'memused': [435.92, 435.90, 436.02, 436.02, 436.19]}
df = pd.DataFrame(raw_data)
df['time_c'] = df['time'].cumsum().round(2)
# Plotting code.
data = []
layout = {'margin': {'t': 105},
'title': {'text': 'Example Showing use of Secondary X-Axis',
'y': 0.97}}
# Create a (hidden) trace for the xaxis.
data.append({'x': df.index,
'y': df['memused'],
'showlegend': False,
'mode': 'markers',
'marker': {'size': 0.001}})
# Create the visible trace for xaxis2.
data.append({'x': df['time_c'],
'y': df['memused'],
'xaxis': 'x2',
'name': 'Inference'})
# Configure graph layout.
nticks = int(df.shape[0] // (df.shape[0] * 0.05))
layout['xaxis'] = {'title': 'Number of Inferences',
'nticks': nticks,
'range': [df.index.min(), df.index.max()],
'tickangle': 45,
'type': 'category'}
layout['xaxis2'] = {'title': 'Time(ms)',
'nticks': nticks,
'overlaying': 'x1',
'range': [df['time_c'].min(), df['time_c'].max()],
'side': 'top',
'tickangle': 45,
'type': 'category'}
layout['yaxis'] = {'title': 'Memory Used (MB)'}
fig = {'data': data, 'layout': layout}
plot(fig, filename='/path/to/graph.html')
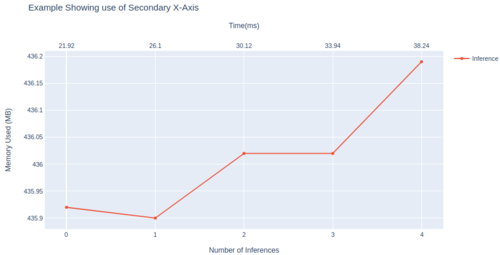
示例图(原始数据集):
为了代码简单起见,我故意省略了任何额外的显示配置。然而,参考顶级的plotly 文档,这些图表是高度可配置的。
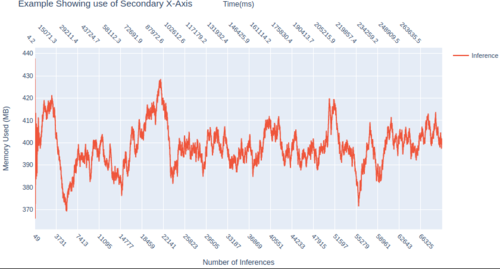
示例图(新数据集):
该图使用来自其他答案的(更大,70k 行)合成数据集。
TA贡献1909条经验 获得超7个赞
尽管通常不鼓励,但我将发布另一个答案来解决新数据集,因为前面的答案在给定原始数据集的情况下有效。
此示例与辅助 x 轴的原始请求不同,原因有两个:
由于(新)数据集的大小,绘制“隐藏”数据层并不是最佳选择。
为了使辅助 x 轴正确显示,必须绘制第二个趋势,并且考虑到之前的原因,这不再是一个选项。
因此,采取了不同的方法——x轴的组合标记。单个 x 轴没有绘制两个轴,而是具有两个所需的标签。
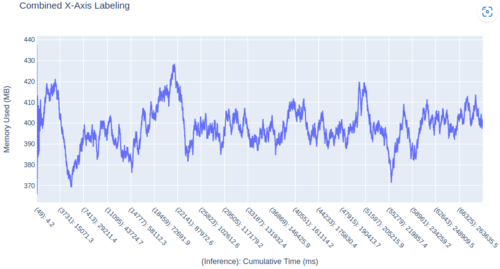
示例图:
注意:这(显然)是合成数据,以便达到更新问题中的行数 (70k)。
示例代码:
import numpy as np
import pandas as pd
from plotly.offline import plot
# Synthesised dataset. (This code can be ignored.)
np.random.seed(0)
a = np.random.exponential(size=70000)*4
t = pd.Series(a).rolling(window=2000, min_periods=50).mean().to_numpy()
r = np.arange(70000).astype(str)
m = t*100
df = pd.DataFrame({'run': r,
'time': t,
'memused': m}).dropna()
# Add cumulative time column.
df['time_c'] = df['time'].cumsum().round(1)
# --- Graphing code starts here ---
def create_labels(x):
"""Function to create xaxis labels."""
return f"({x['run']}): {x['time_c']}"
# Create xaxis labels.
df['xaxis'] = df.apply(create_labels, axis=1)
# Create the graph.
data = []
layout = {'title': 'Combined X-Axis Labeling'}
data.append({'x': df['xaxis'],
'y': df['memused']})
layout['xaxis'] = {'title': '(Inference): Cumulative Time (ms)',
'type': 'category',
'nticks': df.shape[0] // 3500,
'tickangle': 45}
layout['yaxis'] = {'title': 'Memory Used (MB)'}
fig = {'data': data, 'layout': layout}
plot(fig, filename='/path/to/graph.html')
添加回答
举报
