对下述分析方法感兴趣或者没有研究思路的小伙伴,欢迎踊跃探讨!
今天小编分享一篇2022年9月发表在Front Endocrinol (Lausanne)的文章(IF :6.055),题目为“Identification of hub genes for early detection of bone metastasis in breast cancer”。
一.背景&方法
背景:
在全球范围内,在所有妇女中,最常被发现和诊断的、最致命的癌症类型是乳腺癌(BC)。特别是,骨是乳腺癌患者中最常见的远处转移之一,大约80%的晚期患者会发生骨转移。因此,我们需要识别和验证能够区分转移性乳腺癌和非转移性乳腺癌的早期检测标志物。
方法:
1、GSE55715、GSE103357和GSE146661基因表达谱数据从GEO数据库下载
2、GEO2R用于筛选差异表达基因(DEGs)。
3、火山图、维恩图和热图是通过使用ggplot2包生成的。
4、利用R包clusterProfiler对DEGs进行KEGG和GO富集分析。
5、利用STRING数据库构建PPI网络,利用cytoHubba软件识别hub基因。
生成K-M生存曲线和ROC曲线,验证hub基因的表达。
二.结果
1、BC中DEGs的鉴定
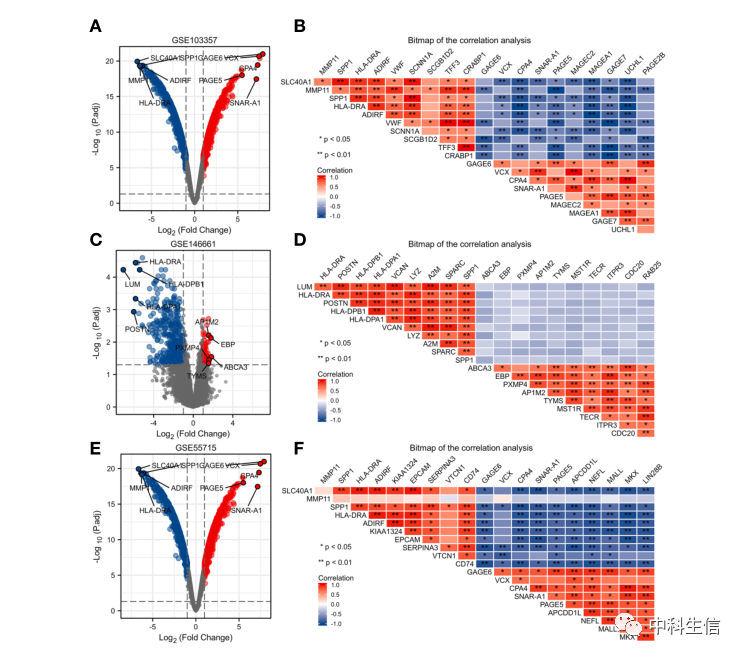
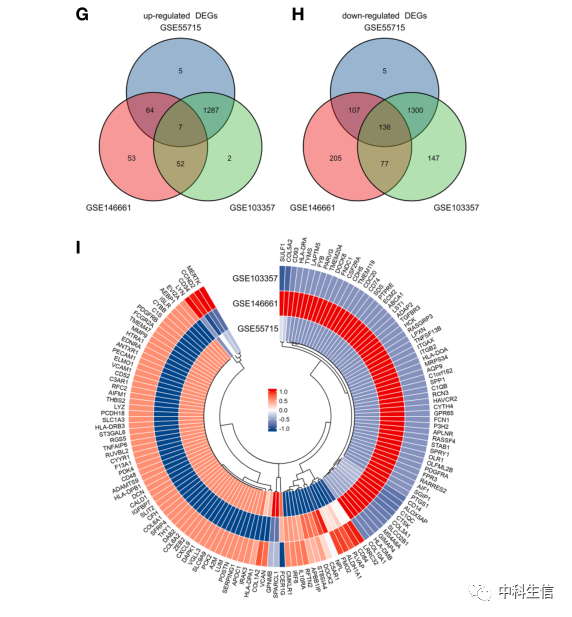
GSE103357、GSE146661和GSE55715中DEGs的鉴定(图1)。(A)显示GSE103357中所有表达基因的火山图。(B) GSE103357中前10个上调或下调的DEG的相关性分析热图。©显示GSE146661中所有表达基因的火山图。(D) GSE146661中前10个上调或下调的DEG之间的相关性分析热图。(E)显示GSE55715中所有表达基因的火山图。(F) GSE55715中前10个上调或下调的DEG的相关性分析热图。(G, H)上调和下调的DEG的维恩图。(I) 143个重叠DEG的环形热图。
2、DEGs的KEGG和GO富集分析
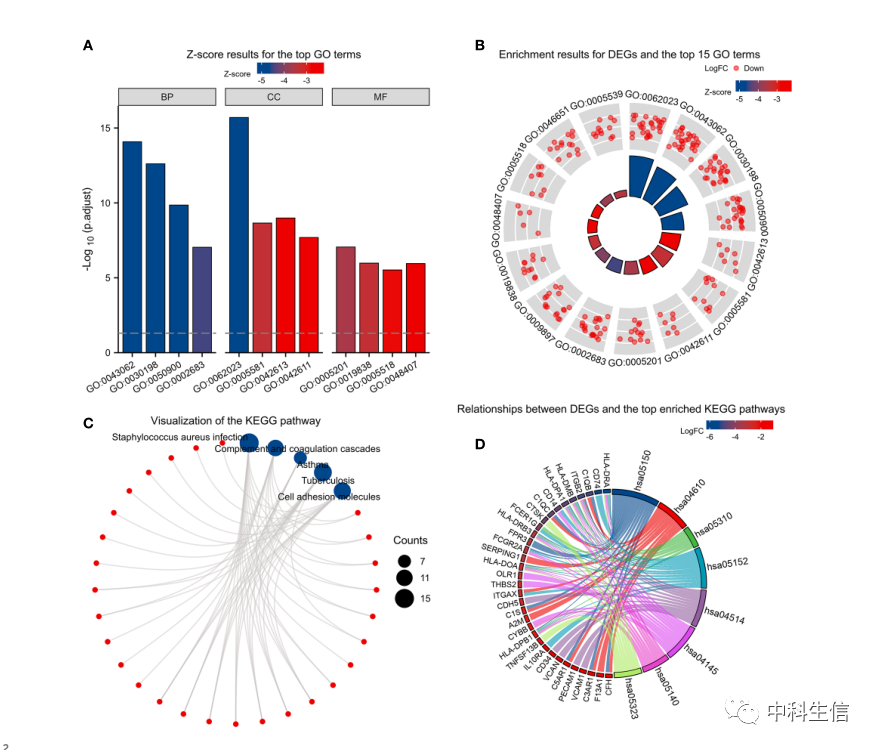
DEGs的KEGG和GO富集分析(图2)。(A)前12个GO术语的z得分结果,包括前4个bp、cc和mf。(B) DEGs和前15个GO富集结果。(C, D) KEGG富集分析。
3、PPI网络构建及hub基因分析
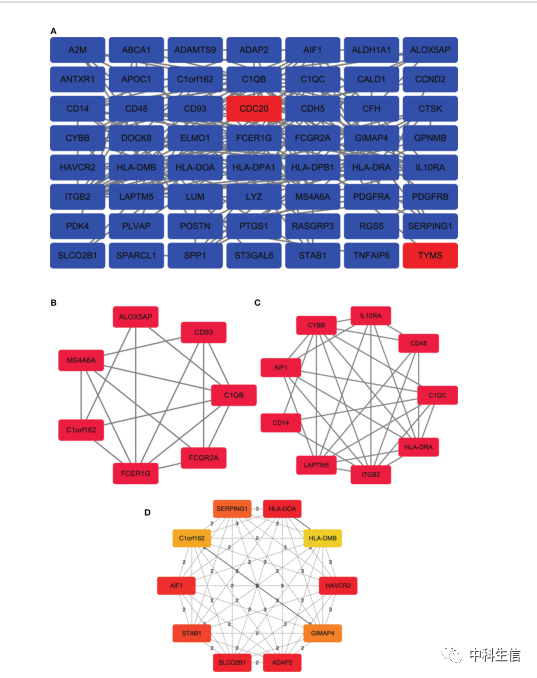
PPI网络构建及hub基因分析(图3)。(A)从56个节点和178条边的PPI网络中获得最显著的模块。(B)模块1包含7个上调基因节点和16个边,MCODE评分=5.33。©模块2包含9个基因节点和28个边,MCODE评分=7。(D)利用cytoHubba插件从PPI网络中选择hub基因。
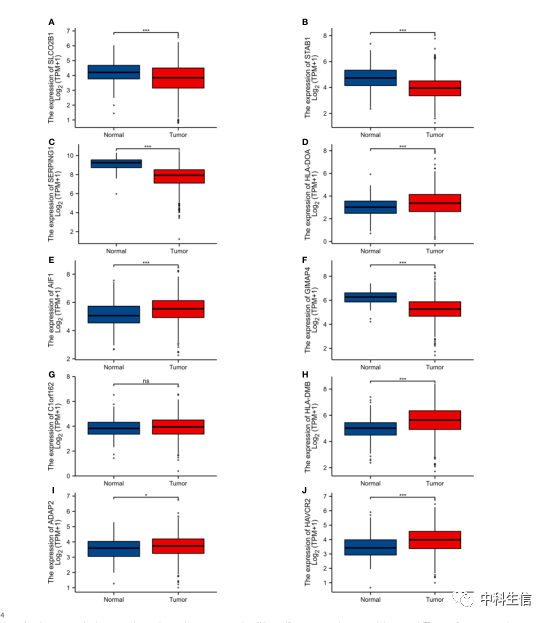
4、BC患者的Hub基因表达
正常样品和肿瘤样品中各种基因的表达(图4)。
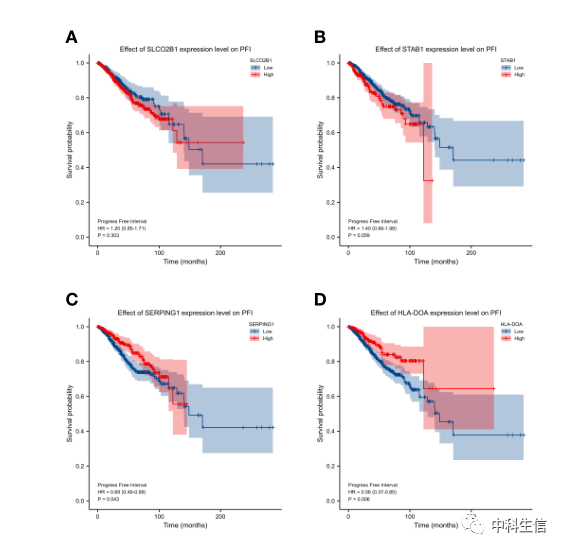
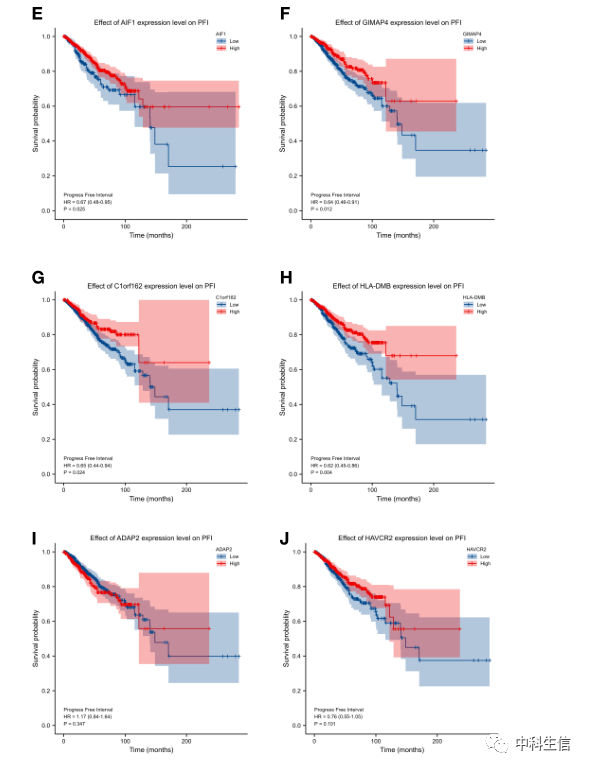
5、hub基因的生存分析
10个hub基因的无进展间隔(PFI)曲线。BC患者各基因高、低表达的无进展间隔(PFI)曲线(图5)。
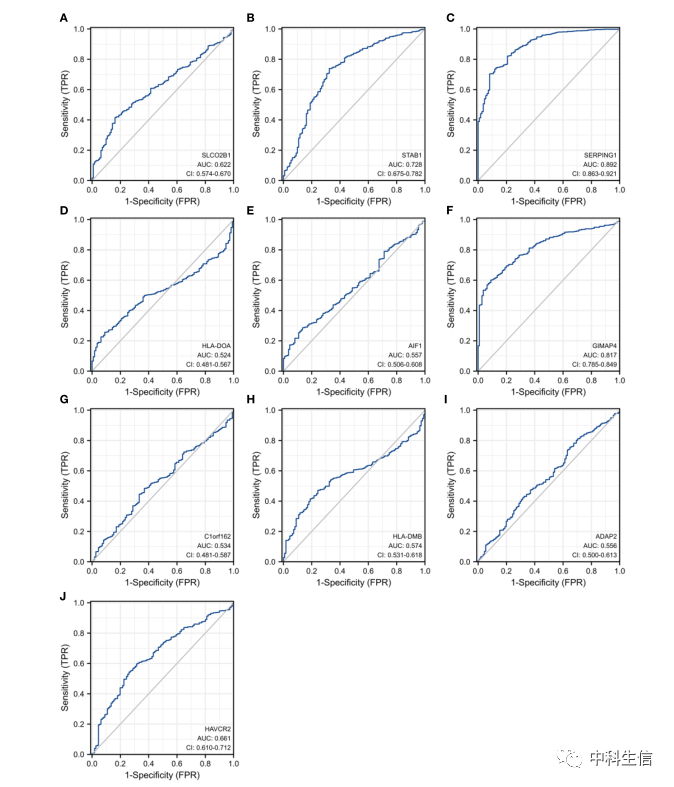
10个hub基因的ROC曲线(图6)。
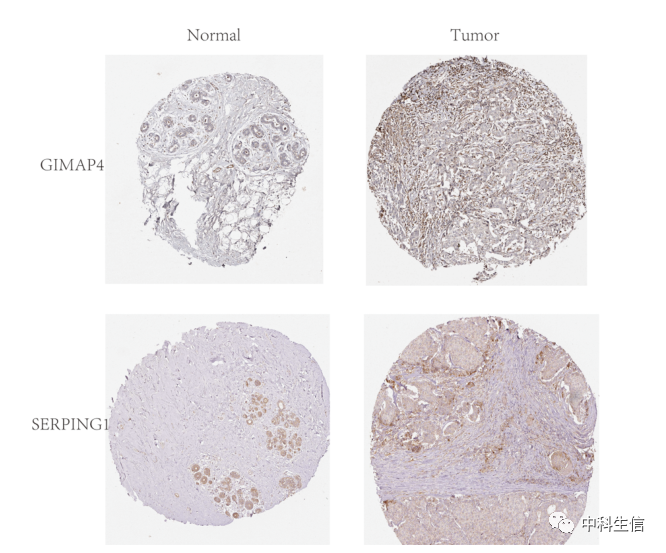
代表性组织学图像来自人类蛋白质图谱数据库(THPA, https://www.proteinatlas.org/)(图7)。
对上述分析方法感兴趣或者没有研究思路的小伙伴,欢迎踊跃探讨!
共同学习,写下你的评论
评论加载中...
作者其他优质文章